Angesichts kontinuierlich wachsender Datenmengen müssen sich Biowissenschaft und Medizin stärker als je zuvor mit dem Thema Data Science auseinandersetzen. Natural Language Processing (NLP) und Natural Language Understanding (NLU) nehmen beim Wissensmanagement eine führende Rolle ein. Wie sonst lässt sich eine öffentliche Literaturdatenbank wie PubMed mit mehr als mehr als 33 Millionen wissenschaftlichen Abstracts proaktiv nutzen?
Verfahren zur computergesteuerten Verarbeitung von Texten und gesprochener Sprache lassen sich unter dem Oberbegriff NLP zusammenfassen. Der Teilbereich NLU bezeichnet das inhaltliche Verstehen von Texten und Sprache. So lassen sich Assoziationen und Zusammenhänge zwischen Wörtern begreifen und interpretieren – also z. B. die Zusammengehörigkeit des Wortes „Blutgruppe“ mit „Transfusion“.
Smarte Datenbanken für NLU: Lesende Helferlein
Damit ein Computer Texte „verstehen” kann, muss das System zunächst dazu fähig sein, einen Satz in seine Struktur zu zerlegen und die richtige Verbindung zwischen Subjekt, Objekt, etc. (Parsing) herzustellen. Dabei wird jedes Wort der jeweiligen Definition im Wörterbuch zugeordnet (Word Sense Disambiguation). Wörter sind wie in einem Lexikon miteinander verknüpft, so dass auch Synonyme, Fremdsprachen und sogar Rechtschreibfehler miteinfließen. Doch die Frage bleibt: Wie lässt sich die Syntax einer Sprache in ein Datenmodell überführen?
Mit herkömmlichen Datenbanken ist dieser komplexe Prozess nicht möglich. Graphtechnologie bietet hier einen Lösungsansatz, durch den Wissenschaftler heterogene Datenmengen in ein ganzheitliches Gesamtbild bringen können. Die Funktionsweise von Graphdatenbanken lässt sich schnell erläutern: Daten werden als Kreise (Knoten) dargestellt, die über Linien (Kanten) miteinander verbunden sind. Diesen können jeweils qualitative oder quantitative Eigenschaften zugeordnet werden. Über das Knoten-Kanten-Prinzip lässt sich das Datenmodell beliebig erweitern – das ermöglicht auch die wirklichkeitsnahe Abbildung großer und komplexer Netzwerke.
Graphen und NLU – ein effektives Forschungsteam
Das einfache Datenmodell lässt sich auf unterschiedlichste Anwendungsfälle übertragen. Ein Graph in der medizinischen Forschung z. B. verknüpft sämtliche spezifische Fachbegriffe, Namen von Erregern, Kürzel, Synonyme, Publikationen in Fremdsprachen und Sachnummern im Graphen. Kombiniert man NLU mit Graphtechnologie, spricht man von NLU-Knowledge Graphen. Diese eignen sich ideal zur Sprachverarbeitung und für Machine Learning in wissensbasierten Unternehmen.
NLU-Algorithmen kommen zum Einsatz, um Schlüsselwörter in Texten zu identifizieren und relevante Absätze, Phrasen und Sätze zu erkennen und zu extrahieren. So kann „zwischen den Zeilen“ gelesen werden. Theoretisch lassen sich Publikationen nach bestimmten Kriterien bewerten, priorisieren, filtern und gezielt mit anderen gegenüberstellen. So ist es möglich, Rankings zu erstellen. Zusammenhänge zwischen Langzeitstudien werden sichtbar. Damit haben Wissenschaftler zu jedem Zeitpunkt einen ganzheitlichen Blick auf den aktuellen Forschungsstand.
Im Graphen lässt sich der gesamte Wissensschatz verknüpfen und durchsuchen. Einmal getätigte Abfragen werden gespeichert und können für zukünftige Interaktionen erneut herangezogen werden. Der Knowledge Graph lebt, wächst und entwickelt sich ständig weiter – so wie die Sprache und die Texte auch. Es entsteht ein semantischer Wissenskontext, in dem Wissenschaftler Strukturen erkennen, die auf den ersten oder zweiten Blick nicht offensichtlich sind. Data Scientists bewegen sich frei in alle Richtungen und folgen Verknüpfungen über mehrere Knoten hinweg, tauchen in Datengruppen ein und haben dabei den vollen Zugriff auf die neusten biomedizinischen Daten.
Wie effizient NLU-Knowledge Graphen arbeiten und wie genau sich die medizinische Forschung diese Technologie zu Nutze macht, zeigen die folgenden Beispiele.
Knowledge Graphen gegen Magenkrebs
Ein amerikanisches Molekulardiagnostikunternehmen nutzt Graphtechnologie bei der Erforschung gastrointestinaler Tumore (Magenkrebs) und entwickelte eine digitale Mikrofluidik-Plattform, die es Wissenschaftlern ermöglicht, Krankheiten anhand von microRNAs nachzuweisen. Um microRNA-Biomarker zu finden, mussten die Forscher Erkenntnisse aus umfangreicher medizinischer Forschung zunächst assimilieren und in einem Knowledge Graphen erfassen.
Beim Extrahieren von Informationen aus der medizinische Zentralbibliothek PubMed ging das Team schrittweise vor. Mit Hilfe des freien Frameworks Apache Hadoop wurden zunächst alle relevanten Artikel (rund 1 Milliarde) zum Thema mRNA „gelesen“ und die Texte in Sätze und Daten zerlegt. Das Ergebnis war ein Datensatz aus insgesamt 200.000 Millionen Sätzen und 100.000 Millionen Concept Mentions. Auf dieser Basis trainierte das Team im nächsten Schritt ein Machine Learning (ML)-Model, um die Beziehungen zwischen den Entitäten bzw. Textbausteinen zu erkennen und zu klassifizieren. Erst dann wurden die neuen Informationen – einschließlich des ML-Modells – in die Graphdatenbank überführt. Dort erlaubt die farbcodierte interaktive Visualisierung eine intuitive Suche. Mit einem Klick lässt sich der gesamte Artikel sowie dazugehörige Studien einsehen.
Das DZD: Graphtechnologie für effiziente Diabetesforschung
Auch in der Diabetesforschung stoßen Forscher beim Lesen und Verarbeiten von Fachpublikationen schnell an ihre Grenzen. Das Deutsche Zentrum für Diabetesforschung, kurz DZD, hat sich die Erforschung und Entwicklung innovativer Strategien zur Prävention, Früherkennung und Behandlung von Menschen mit Prädiabetes oder Diabetes auf die Fahne geschrieben. Um unvernetzte Datensilos in den Griff zu bekommen, baute das DZD ein standortübergreifendes Daten- und Knowledge Management auf: DZDconnect.
Auch dieses System basiert auf einer Graphdatenbank und erstellt Verknüpfungen zwischen Metadaten aus Grundlagenforschung und klinischen Studien. Diese Daten sollten um disziplinübergreifende Forschungsdaten aus öffentlichen Quellen erweitert werden. Dazu gehören auch Daten zu anderen relevanten Krankheiten (z. B. Schlaganfall, Herzinfarkt, Krebs und Alzheimer) sowie hochstandardisierte Daten aus Tiermodellen (z. B. Mäuse). Diese Verknüpfung erlaubt es, Rückschlüsse auf das humane System zu ziehen und Ähnlichkeiten bei einzelnen Genen und Stoffwechselvorgängen zu untersuchen. Zum Beispiel: Welcher Diabetes-Typ lässt sich auf welche Gene zurückführen? Wie wirken sich externe Faktoren aus?
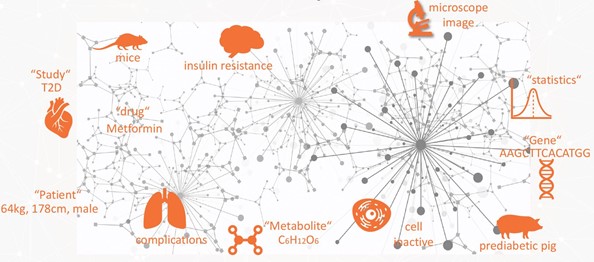
Insgesamt galt es, 30 Mio. Veröffentlichungen aus der PubMed in die Forschungsarbeit miteinzubeziehen. Im Schnitt brauchte das DZD rund 1,5 Sekunden, um einen Abstract zu analysieren. Was auf den ersten Blick schnell erscheint, mündet bei der Menge an vorliegenden Forschungsdaten in rund eineinhalb Jahren Analyse-Arbeit. Der Ansatz von NLP und Graphdatenbank des DZD läuft daher parallel und automatisiert im Hintergrund ab. Ausgeklügelte Algorithmen führen eine semantische Analyse der Texte durch, erkennen relevante Entitäten und verknüpfen sie mit internen Informationen in der Datenbank. So betrachtet das DZD medizinische Fragen ganzheitlich und arbeitet indikationsübergreifend.
Knoten und Kanten für beschleunigte Medikamentenentwicklung
Vor einer ähnlichen Herausforderung der Datenverarbeitung stand auch ein Schweizer Biotechnologie- und Pharmaunternehmen. Rund 1 Mrd. historische Datensätze aus jahrzehntelanger Grundlagenforschung sollten mit den Daten aus großen Bibliotheken wie PubMed verknüpft werden. Auch hier kommt NLP/NLU zusammen mit Graphtechnologie ins Spiel.
Mithilfe von Text Mining und Association Rule Mining gelang es den Forschern, alle verfügbaren Informationen in einem Knowledge Graphen zu bündeln. Das Team erstellte eine Pipeline, um Texte in über 5.600 wissenschaftlichen Zeitschriften zu scannen und relevante Information aus PubMed zu extrahieren. Um in der Literatur-Metadatenbank nach relevanten Schlagwörtern und spezifischen Themen zu suchen, griffen die Biotechnologen auf die sogenannten MeSH-Terms zurück. MeSH ist eine in PubMed hinterlegte Schlagwortliste, die jeden indizierten Artikel mit 10-12 MeSH-Begriffen versieht. Bei der Suche mit MeSH-Begriffen wird nicht im Freitext, sondern nach Vorkommen von Wörtern in den Referenzen gesucht. Dabei bezieht die Suche automatisch Wortvarianten, Wortendungen, Plural- oder Singularformen sowie Synonyme automatisch mit ein.
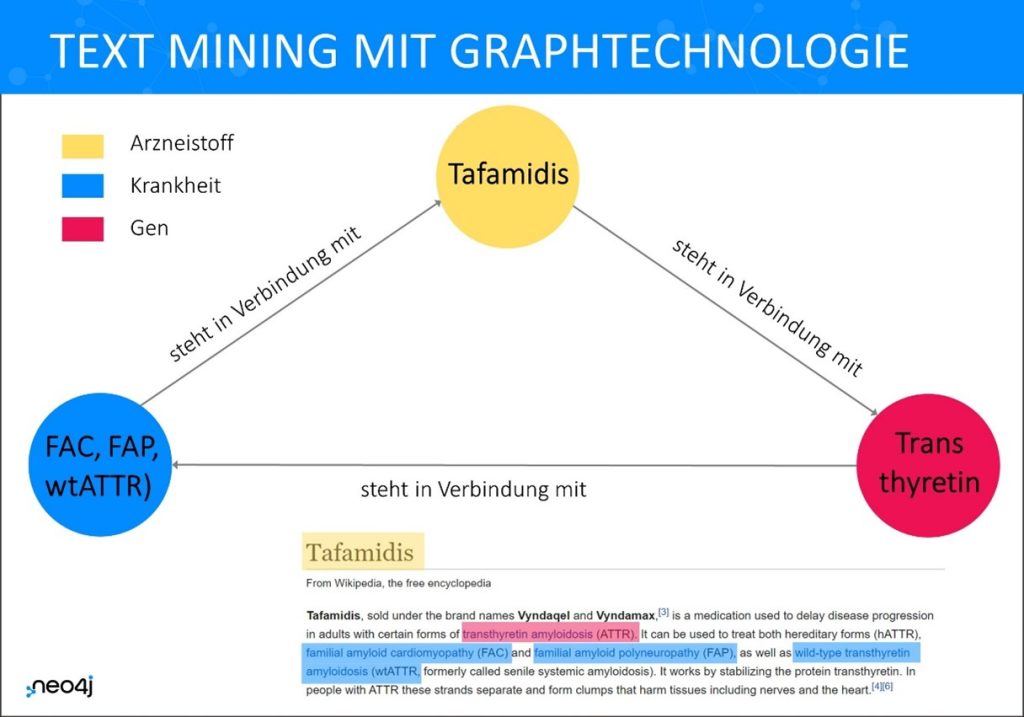
Die extrahierten Daten wurden anschließend gemeinsam mit historischen Daten und Bilddaten des Schweizer Unternehmens in die Graphendatenbank eingespeist. Zur Kodierung des Datenmodells definierten die Forscher 15 Knoten-Kategorien sowie 90 mögliche Beziehungen zwischen den Knoten. Anhand dieses Bauplans wurden schließlich die Beziehungen im Dreieck aus Krankheit-Target-Wirkstoff im Graphen modelliert. Das biologischen Gesamtwissen des Schweizer Unternehmens in einer ganzheitlichen Übersicht ermöglicht den Forscherteams volle Bewegungsfreiheit und hilft letztendlich, Medikamente schneller auf den Weg zu bringen.
In allen drei Fallbeispielen bauen Wissenschaftler auf vorhandenes Know-How und nutzen gleichzeitig das externe Wissen der Forscher-Community. Die smarten Lösungen zum Wissensmanagement basieren auf neuen Technologien wie NLP/NLU, ML und Graphtechnologie. So lässt sich der Lese-Last in der medizinischen Forschung ein effektives Tool entgegensetzen.



Um einen Kommentar zu hinterlassen müssen sie Autor sein, oder mit Ihrem LinkedIn Account eingeloggt sein.